Científicos descubren tres bacterias que no eran diagnosticadas y que podrían enfermar a las personas

La investigación es un proyecto inscrito en el CIET-UCR, en el cual participaron once científicos y científicas. Ocho son de la UCR, dos alemanes y uno de la Universidad de Hokkaido, Japón. De izquierda a derecha, en la fotografía están el Dr. César Rodríguez Sánchez, el Dr. Carlos Quesada Gómez y el Dr. Esteban Chaves Olarte.
Investigadores de la Facultad de Microbiología de la Universidad de Costa Rica (UCR) descubrieron en el país la existencia de tres nuevas especies de bacterias (nunca antes descritas) que son “hermanas mayores” del perjudicial patógeno Clostridium difficile, microorganismo que causa serios cuadros de diarrea y que puede llevar a la muerte si no es tratada a tiempo.
Así lo informó el Dr. César Rodríguez Sánchez, director del Centro de Investigación en Estudios Tropicales (CIET-UCR), y cuyos hallazgos se encuentran publicados en la revista científica Scientific Reports, de la casa editorial Nature, y en la revista eLife; ambas consideradas de alto prestigio internacional.
Recordemos que el C. difficile es una de las causas más importantes de diarrea hospitalaria en el mundo, generada, principalmente, por el uso excesivo de antibióticos durante periodos prolongados. En el 2008, el Hospital San Juan de Dios sufrió un brote por este patógeno que alcanzó su pico máximo en abril del 2009. Incluso, se llegó a documentar un exceso de 216 casos sobre lo esperado, según constata una publicación científica en el Acta Médica Costarricense.
En ese momento, el sistema sanitario nacional no tenía la infraestructura ni la experiencia para poder hacer análisis altamente especializados de Clostridium difficile, por lo que la UCR se encargó de apoyar a la Caja Costarricense de Seguro Social (CCSS) y de confirmar los diagnósticos, así como la identidad de las cepas circulantes. Lo anterior, como parte de un proyecto de acción social coordinado por el Laboratorio de Investigación en Bacteriología Anaerobia (LIBA-UCR).
Ahora, después de años de trabajar en conjunto, se cosechan nuevos hitos científicos. Los investigadores de la UCR no se quedaron solamente en emitir los diagnósticos de laboratorio a las y los pacientes, sino que también analizaron por qué algunas muestras enviadas por la CCSS, que generaban un cuadro clínico similar al producido por el C. difficile, estaban escapando de las pruebas diagnósticas.
Dichos estudios finalmente se concretan al revelar el motivo y decirle al mundo, por primera vez, que se debe a la existencia de tres nuevas especies de bacterias que son familia cercana del Clostridium difficile y las cuales, incluso, hasta ya fueron profundizadas por entidades internacionales, incluida la Universidad de Harvard.
“De nuestro vínculo con la Caja, en algún momento comenzaron a salir cepas que no cumplían con las características originales y que nos llamaban la atención. Inicialmente, pensamos que escapaban de nuestros métodos. Sin embargo, después de repetirlos en varias ocasiones, nos dimos cuenta de que las cepas tenían que ser diferentes. Entonces, secuenciamos su genoma y nos percatamos que, efectivamente, eran tan diferentes que ni siquiera se podrían considerar Clostridium difficile, sino que eran familia y que eran remitidas porque causaban un cuadro clínico similar”, comentó el Dr. Rodríguez.
El descubrimiento proporciona un panorama pionero en Costa Rica. Las tres bacterias fueron encontradas en dos hospitales clase A del país, y al momento de la investigación, las y los científicos solo conocían reportes que indicaban la existencia de única especie cercana al Clostridium, hecho que cambió con el estudio costarricense.
“Cuando nosotros venimos y hacemos los análisis, resulta que en nuestras manos no tenemos solo una, sino otras tres más. Por lo tanto, venimos a contribuir a demostrar que la diversidad de esas especies cercanas al Clostridium era mayor de lo que se pensaba, porque existía el reporte de una cuando, realmente, existían otras tres más que hallamos aquí, en Costa Rica”, amplió el Dr. Rodríguez.
Para el Dr. Carlos Quesada, microbiólogo del LIBA-UCR y cómplice en el descubrimiento, que esto fuera posible gracias a un proyecto de acción social retrata no solo la vital importancia de este pilar, al brindar diagnósticos que jamás fueron cobrados a la CCSS por el bienestar del país, sino que también brinda insumos a la investigación científica.
“Este proyecto de acción social ha funcionado como una plataforma para proporcionar distintas cepas de Clostridium difficile a los proyectos de investigación que se han ejecutado, principalmente, en el CIET-UCR. Así, se dio un engranaje muy interesante en dos pilares de la UCR. En este tiempo se ha logrado recolectar esas tres cepas y muchísimas otras más que han permitido desarrollar proyectos de investigación relacionados con el C. difficile, por ejemplo: epidemiología molecular, patogénesis, resistencia a los antibióticos”, manifestó el Dr. Quesada.
En efecto. Desde la perspectiva del Dr. Esteban Chaves Olarte, otro de los científicos partícipes, justamente ese trabajo es el que hoy permite advertirle al país que los sistemas diagnósticos actuales, de alguna manera, pueden ser ciegos a ciertos eventos producidos por cepas de Clostridium difficile. “Esto, por un lado, obliga a mejorar los sistemas diagnósticos, lo que constituye un aporte fundamental en el campo de la microbiología diagnóstica y una incursión en el campo biotecnológico potencialmente patentable”, afirmó.

Mediante el proyecto de acción social ED-239: “Laboratorio de anaerobios al servicio de la comunidad”, la UCR ha logrado consolidar un cepario con más 1 000 especies diferentes de C. difficile durante casi una década. Así, se le da al país algo que nadie más hace: mejores diagnósticos sin cobrarle a la CCSS e investigación de vanguardia.
Foto: Anel.Las tres especies recién identificadas son raras. En la colección mundial de aislamientos que han sido secuenciadas, se tiene pocos genomas de las nuevas y 100 000 de las C. difficile tradicional. Esto es una muestra clara que se está ante la punta de un iceberg donde hay muchas otras especies diferentes a C. difficile cercanas y que también comparten las mismas toxinas.
¿Más perjudiciales?
¿Y qué se sabe de esas tres nuevas especies? ¿Estamos ante microorganismos más perjudiciales para el ser humano? La respuesta es solo una: se sabe poco.
Por el momento, se conoce que podrían enfermar a las personas, pero no qué tan virulentas o peligrosas para la salud son estas tres especies, pues apenas se acaban de identificar.
Lo poco que se conoce (desde una perspectiva genética) es que ellas provienen del mismo antecesor común que, si bien no le dieron origen a la actual C. difficile, sí divergieron del mismo “padre” hace millones de años. Así que las especies recién descubiertas son familiares más viejas que la misma C. difficile.
“Es importante que quede claro que Clostridium difficile (que causa mucho problema a nivel hospitalario y que lo conocemos como un patógeno nosocomial) no se deriva de las especies recién descubiertas, sino que tanto C. difficile, como las nuevas provienen de un mismo ancestro extinto, pero que se separaron en algún momento y que estas nuevas se dividieron antes que Clostridium difficile”, puntualizó el Dr. Rodríguez.
Para el especialista, lo anterior es asombroso, pues la rama evolutiva de estos patógenos emergentes se conoció al revés de lo que normalmente se da de forma cronológica. Es decir, primero se hallaron los últimos que evolucionaron y, al estudiar más en profundidad, salieron los antecesores, que no son el origen de C. difficile, pero sí familia.
“Todavía no sabemos por qué las especies encontradas saltaron al ser humano y la razón se debe a la cantidad de aislamientos. Las nuevas cepas cercanas a difficile que existen se cuentan en los cientos, mientras que las cepas que conocemos de Clostridium difficile se cuentan por decenas de miles. Esa asimetría tan grande hace que no sea posible hacer una comparación científicamente válida en este momento para saber qué fue lo que realmente le permitió a las especies recién descritas entrar al ser humano”, afirmó el Dr. Rodríguez.
Lo que sí se sabe, aseveró ese mismo científico, es que la diversidad, tanto de los genomas como de las toxinas en las especies hermanas de Clostridium difficile, es inusualmente alta. Por eso, tan solo una fracción de esa gran pluralidad que se ve en el ambiente es la que se observa en las cepas que infectan a humanos.
Es como si de una gran piscina de pelotas plásticas para niños, en la cual las pelotas son bacterias, solo algunas lograrán salir, adaptarse al ser humano e iniciar su propagación.
"No se sabe cómo las nuevas especies proliferan ni su hospedero. Se cree que el humano es accidental. Por lo tanto, el hallazgo se vuelve muy interesante en el estudio de pandemias para saber qué ocurre y por qué un microorganismo, principalmente ambiental, terminó en el humano. Es vital recordar que la salud humana no solamente depende de los hospitales, sino también del ambiente".
Dr. César Rodríguez Sánchez.
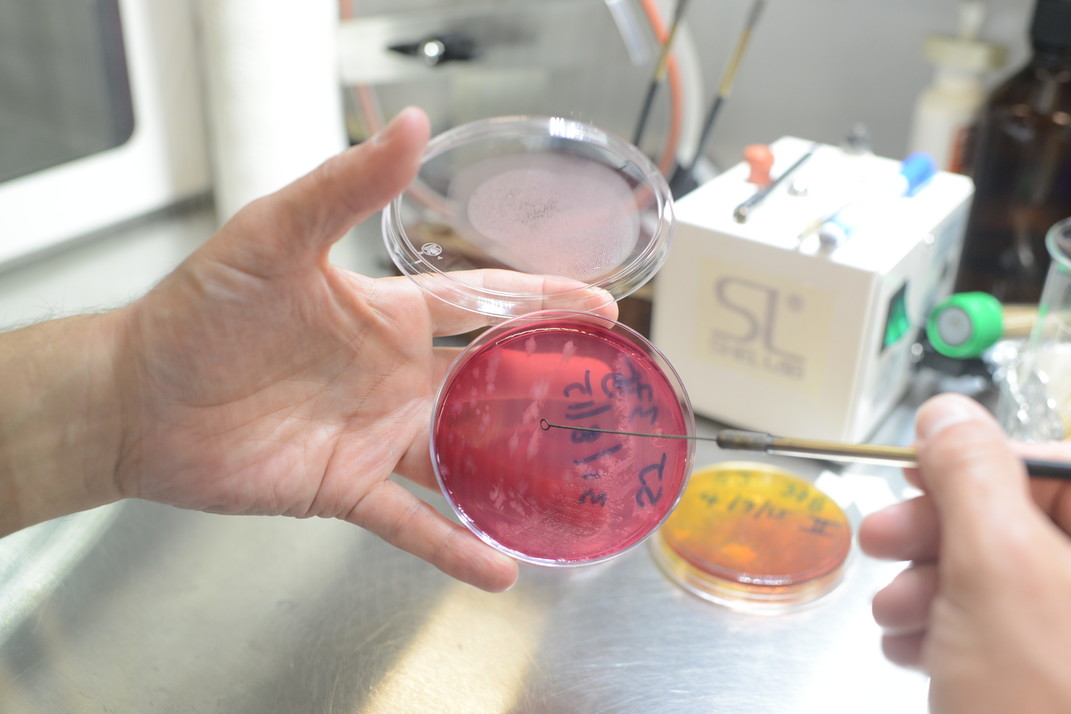
Los tres nuevos patógenos se han encontrado en hospitales clase A. En total, desde el inicio del proyecto de acción social se han analizado aproximadamente entre 1 500 a 2 000 muestras bajo sospecha de C. difficile. De esas, tan solo siete han dado lugar a estas nuevas especies.
Foto: Anel.Impacto nacional
La pregunta ahora es, ¿cómo ayudan esos descubrimientos a la salud nacional? Para el Dr. Esteban Chaves Olarte un aporte está en contribuir en mejores diagnósticos y así evitar subregistros.
Por mucho tiempo, el utilizar solo pruebas de laboratorio orientadas a identificar el C. difficile en humanos no ha sido efectivo para detectar nuevas especies. Además, hace creer que las personas enfermas con la diarrea severa tienen C. difficile cuando, realmente, tienen otros patógenos.
De esta manera, la contribución gestada desde la UCR da insumos para mejorar los diagnósticos a nivel nacional e internacional, al tener más certeza científica de que la carencia de un marcador en particular es un posible indicador de estar ante una de estas cepas hermanas al C. difficile.
“Estos aportes permiten mejorar y complementar las técnicas diagnósticas actuales y, adicionalmente, contribuir mediante estrategias alternativas. La comunicación de estos descubrimientos a la comunidad médica genera una alerta para aquellos casos muy sugestivos de enfermedades producidas por el Clostridium difficile, pero que brinden resultados negativos. Para estos casos, el equipo de investigación de la UCR está en la capacidad de brindar alternativas diagnósticas, como la prueba de citotoxicidad celular o el cultivo de la bacteria, que contribuiría en forma directa al bienestar de los pacientes afectados”, fundamentó el Dr. Chaves.
Adicional a ese aporte, el Dr. Carlos Quesada agregó que esa nueva información también coopera en los estudios de resistencia de los antibióticos. Esto es vital. A nivel mundial y nacional, cada vez es más frecuente el uso de antibióticos para tratar diversas infecciones. Muchas de ellas, incluso, ya son resistentes a los tratamientos actuales.
Eso genera un efecto colateral importante: entre más infecciones hay, más antibióticos de diferente tipo se deben usar en casos de resistencia, lo cual, a su vez, lleva a un desequilibrio perjudicial de las bacterias buenas que necesita el intestino para funcionar correctamente. A esto se le conoce como disbiosis intestinal y es el estado perfecto para que el C. difficile prolifere.
Como si eso no fuera suficiente, en ese campo de resistencia a los antibióticos el Dr. Quesada aseguró que la UCR ha localizado que algunas cepas de C. difficile ya presentan resistencia al linezolid, un antibiótico muy fuerte. Aunque el linezolid no es un tratamiento de primera elección contra el C. difficile, sí es muy utilizado para abordar las infecciones complicadas de otras bacterias. Es decir, sin querer el C. difficile funciona como reservorio de un mecanismo de resistencia al linezolid.
“Estos trabajos sobre la resistencia a los antibióticos en los diferentes aislamientos del C. difficile obtenidos permiten proporcionar información a fin de mejorar el uso de estos fármacos dentro del hospital para otros tipos de infecciones distintas a la del C. difficile. También, ayuda a monitorear la posible resistencia, propiamente del C. difficile, para una mejor toma de decisiones sobre qué tratamiento proporcionar”, argumentó el Dr. Quesada.
Alcance internacional
En el ámbito mundial, los descubrimientos del equipo costarricense no han pasado desapercibidos. Dieciocho científicos internacionales, incluidos de la Universidad de Harvard, solicitaron a la UCR el material biológico para estudiar si estas nuevas cepas ambientales y clínicas cercanas al C. difficile usaban los mismos receptores (sustancias que les permite entrar a la célula humana). Resultó que no.
Dicho hallazgo se divulgó en el 2022 en un artículo científico en la revista Nature Communications. Ahí revelaron que las toxinas de las nuevas especies ni siquiera usan los receptores conocidos para los C. difficiles que infectan a humanos.
De las 206 variantes de la toxina B que estudiaron ―el principal factor de virulencia que le permite a un microorganismo invadir a un huésped― la especificidad de las toxinas se restringen a solo seis u ocho aminoácidos. Cambios en esa pequeña región determina si causa (o no) algún tipo de efecto en la célula y hasta qué tipo de células infecta.
“Cuando los científicos de Harvard se dan cuenta de que es una misma toxina, pero altamente divergente, les surge la inquietud sobre si, biológicamente, se comporta igual que las tradicionales, pero no. Tan solo pequeños cambios en esos aminoácidos pueden dictar que la toxina no pueda actuar sobre el humano. Es como tener un edificio de 15 pisos y que una sola una pequeña ventana defina qué puede o no infectar la toxina”, añadió el Dr. Rodríguez.
Ese resultado implica mucho. Primero, en la presentación clínica y, posteriormente, en sugerir nuevos abordajes terapéuticos.
El Dr. Rodríguez dijo que al conocer los receptores que usa la bacteria para causar enfermedad en el ser humano, en un futuro se podría proponer terapias orientadas a bloquear y evitar que las toxinas se puedan unir a las células. En palabras sencillas, encontrar el talón de Aquiles.
“Todavía no sabemos cuáles presentaciones clínicas genera estas tres especies recién identificadas, porque no hemos tenido la posibilidad de ver en detalle los expedientes clínicos de los pacientes infectados con esas cepas. No obstante, uno puede anticipar que probablemente intoxiquen otros tipos de células a nivel intestinal y que los receptores pueden ser también un blanco terapéutico porque, si se bloquea el receptor, la toxina no puede entrar en la célula y no la va a intoxicar”, detalló el Dr. Rodríguez.
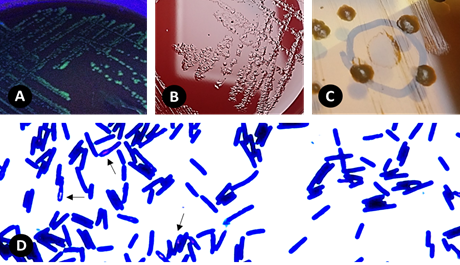
En esta imagen se puede ver el crecimiento de la bacteria. Todas las especies hermanas de C. difficile encontradas en pacientes tienen la toxina B, su principal factor de virulencia. Sin embargo, su toxina, aunque es la misma, es altamente diferente en sus componentes, algo “muy inusual”.
Lo que falta
Hace poco la UCR dio inicio una investigación de maestría en modelos animales y celulares para estudiar la virulencia de estas cepas y compararla con los estudios tradicionales. El objetivo es ver si hay diferencias en la patogenicidad (mecanismo de virulencia), pues se sospecha que las tres tendrían menos capacidad para producir una enfermedad.
Esa hipótesis tendría sentido, pues el C. difficile tradicional le ha tomado millones de años especializarse para afectar al ser humano. Las nuevas no son patógenos humanos, sino especies ambientales, y no han pasado por todo ese proceso de perfección de la estrategia de la virulencia para enfermar a las personas.
Asimismo, los nombres oficiales de las nuevas bacterias todavía están pendientes. El Dr. Rodríguez explicó que las descripciones taxonómicas (obligatorias para clasificar a los microorganismos) tienen un procedimiento.
El proceso implica depositar esas cepas en colecciones internacionales, de forma que el recurso esté disponible para otros investigadores, con el objetivo de que puedan estudiarlas y colaborar. Además, se debe cumplir con una serie de mecanismos legales nacionales e internacionales que están en vías de efectuarse.
No obstante, aunque todavía faltan esas descripciones taxonómicas, la descripción del genoma dada a conocer en el ámbito científico internacional, y ya estudiadas por pares internacionales, las coloca como un hecho científico fuertemente validado.
Revise los artículos científicos aquí
- Revista Scientific Reports. Enlace: https://www.nature.com/articles/s41598-018-32390-6
- Revista eLife. Enlace: https://elifesciences.org/articles/64325
